Unidad 2 Regresión lineal
2.1 Introducción
Una vez presentados los fundamentos de INLA vamos a utilizarlo para trabajar progresivamente desde los modelos más sencillos a los más sofisticados. Empezamos aquí con el modelo de regresión lineal simple, para continuar generalizando con el de regresión lineal múltiple.
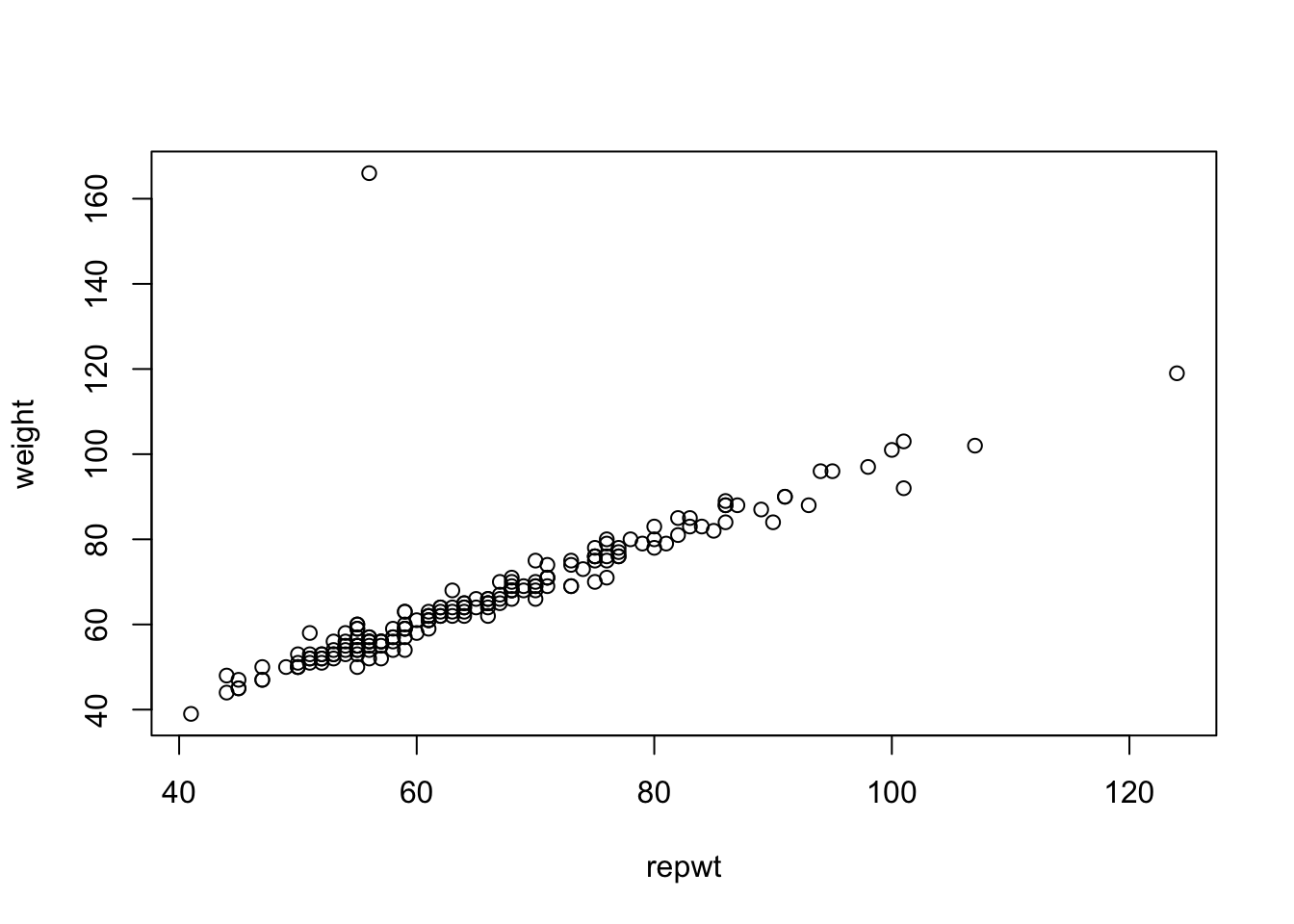
Partimos de la base de datos Davis (en la librería carData), que contiene 200 registros de 5 variables relacionadas con un estudio sobre habituación de hombres y mujeres a la realización de ejercicio físico de forma regular. Las variables que se registraron son sexo, peso y altura (reales y reportados). Vamos a indagar la relación entre el peso real (weight) y el reportado (repwt) a través de un análisis de regresión lineal simple.
2.2 Variables y relaciones
Entendemos como variable respuesta y=weight, de tipo numérico (continua), y como variable explicativa o covariable, x=repwt, también numérica.
La especificación de respuesta \(y\) y predictores \(x_1,x_2,...\), en INLA se registra en una fórmula del tipo:
formula= y ~ 1 + x_1 + x_2 +...en la que podemos prescindir del \(1\) que identifica la interceptación, pues el ajuste, por defecto, siempre se resolverá con su estimación, salvo que en su lugar se escriba un ‘-1’.
En nuestro problema tendríamos pues:
formula = weight ~ repwtA continuación es procedente elegir el modelo sobre la respuesta, o lo que es lo mismo, la verosimilitud.
2.3 Verosimilitud
En principio es razonable asumir normalidad en la respuesta, además de independencia entre todas las observaciones. Así, el modelo propuesto para la respuesta es:
\[(y_i|\mu_i,\sigma^2) \sim N(\mu_i,\sigma^2), i=1,...,n\]
donde y=weight y x=repwt, e \(i=1,...,n\) es un subíndice que identifica a cada uno de los registros disponibles en el banco de datos. La media esperada contiene la relación lineal entre covariable y respuesta, \(\mu_i=\theta+\beta x_i\), donde \(\theta\) es la interceptación de la recta de regresión y \(\beta\) el coeficiente que explica la relación lineal entre \(x\) e \(y\). El vector \((\theta,\beta)\) identifica los efectos latentes, en cuya inferencia posterior estamos interesados, y que están involucrados directamente en la media o predictor lineal. El modelo, o lo que es lo mismo, la verosimilitud, depende también de un parámetro de dispersión \(\sigma^2\) sobre el que también querremos inferir.
Veamos cómo especificar este modelo con INLA. La función names(inla.models()) proporciona un listado de todos los
tipos de modelos posibles, tanto para los datos (likelihood), para los efectos latentes (latent), los parámetros
(prior), y otras opciones que de momento no nos interesan. El listado completo de todas las distribuciones disponibles para cada uno de los tipos de modelos lo obtenemos con el comando inla.list.models(). En particular, si ejecutamos names(inla.models()$likelihood), obtenemos todas las distribuciones disponibles para modelizar los datos.
La distribución gaussian identifica la distribución normal que hemos planteado en nuestro modelo de regresión lineal. Para obtener información sobre cómo está parametrizada y cuáles son las priors por defecto, basta consultar la documentación gaussian con el comando:
# documentación (parametrización y priors) del modelo normal
inla.doc("gaussian")Para ajustar un modelo sencillo en INLA hay que echar mano de la función inla, en la que introducimos en primer lugar la formula, con la relación entre las variables, a continuación el modelo, en el argumento family, y después el banco de datos sobre el que trabajamos. Si no especificamos el argumento family, la función inla interpreta por defecto la opción gaussian, esto es, normalidad para los datos, de modo que podríamos excluir dicha especificación cuando modelizamos datos normales.
# Asumiendo datos normales
fit=inla(formula,family="gaussian", data)
# equivalente a
fit=inla(formula, data)Adelantamos pues un paso más en nuestro problema, añadiendo la verosimilitud normal y la base de datos.
# ajuste del modelo
formula = weight ~ 1+ repwt
fit=inla(formula,family="gaussian",data=davis)2.4 Hiperparámetros
INLA identifica como hiperparámetros todos aquellos parámetros en el modelo que no se corresponden con efectos latentes, esto es, relacionados con el predictor o respuesta esperada. En nuestro modelo, el único hiperparámetro es la varianza \(\sigma^2\), sobre la que es preciso especificar una distribución a priori. Para la varianza \(\sigma^2\) es habitual asumir una gamma inversa difusa, con media y varianza grandes.
En INLA, en lugar de asignar distribuciones a priori sobre las varianzas, se hace sobre el logaritmo de las precisiones, para facilitar el cálculo del máximo de la log-posterior (obtenida de la log-verosimilitud y la log-prior). Así, asumir una gamma inversa difusa \(GaI(\alpha,\beta)\) para la varianza es equivalente a una Gamma difusa \(Ga(\alpha,\beta)\) para la precisión \(\tau=1/\sigma^2\), y una log-gamma difusa \(Log-Gamma(\alpha,\beta)\) para la log-precisión \(log(\tau)\).
Por defecto, como ya verificamos en la documentación de la verosimilitud gausiana, (con inla.doc("gaussian")), la distribución a priori por defecto sobre la log-precisión (\(\theta_1\) en la ayuda) es la log-gamma difusa \(LogGa(1,5\cdot 10^{-5})\), lo que da un valor esperado para la precisión \(\tau\) de 20000 y una varianza de \(4\cdot 10^8\).
Para modificar la distribución a priori de un parámetro podemos utilizar cualquiera de las distribuciones que ofrece INLA en su listado names(inla.models()$prior) (siempre buscando coherencia con la información sobre el parámetro en cuestión).
Para definir una prior para los parámetros o hiperparámetros en INLA hay que definir los siguientes argumentos:
- prior, el nombre de la distribución a priori (para hiperparámetros, alguna de las opciones en
names(inla.models()$prior)) - param, los valores de los parámetros de la prior
- initial, el valor inicial para el hiperparámetro
- fixed, variable booleana para decir si el hiperparámetro es fijo o aleatorio.
La modificación la haremos con el argumento control.family=list(hyper=list(...)) en la función inla, al que le proporcionaremos una lista con el nombre de los parámetros (el short name con el que los identifica INLA en la documentación), que apunta a una lista con la distribución (prior) y los parámetros (param) a utilizar.
En nuestro problema, si tenemos información previa sobre la precisión del modelo, reconocida como prec (short name), y queremos especificar una a priori \(Ga(1,0.001)\) para la precisión, habremos de utilizar la siguiente sintaxis:
prec.info = list(prior="loggamma", param =c(1,0.001))
fit2=inla(formula,family="gaussian",data=davis,
control.family = list(hyper = list(prec = prec.info)))Si nos conformamos con la previa por defecto de INLA, el modelo que estamos asumiendo en nuestro problema de regresión lineal simple será:
\[\begin{eqnarray*} (y_i|\mu_i,\sigma^2) &\sim & N(\mu_i,\sigma^2), i=1,...,n \\ \tau=1/\sigma^2 & \sim & Ga(1,0.00005) \end{eqnarray*}\]
2.5 Efectos fijos
Una variable explicativa entra en el modelo como efecto fijo cuando se piensa que afecta a todas las observaciones del mismo modo, y que su efecto es de interés primario en el estudio.
En un modelo de regresión lineal todos los efectos latentes en el predictor lineal, interceptación y coeficientes de covariables, son efectos fijos. Ante ausencia de información, las priors para los efectos fijos, esto es, (\(\theta,\beta\)) en nuestro caso, se asumen normales centradas en cero y con varianzas grandes. En INLA la interceptación \(\beta\) tiene por defecto una prior gausiana con media y precisión igual a cero (una distribución plana objetiva, que no integra 1), y los coeficientes \(\beta\) también tienen una prior gausiana con media cero y precisión igual a 0.001. Estos valores por defecto se pueden consultar con el comando inla.set.control.fixed.default(), que da la siguiente información:
- mean=0 y mean.intercept=0 son las medias de la distribución normal para los coeficientes \(\beta\) y la interceptación \(\theta\) respectivamente
- prec=0.001 y prec.intercept=0 son las precisiones respectivas de las normales para \(\beta\) y \(\theta\).
Con todo, los parámetros de las priors sobre los efectos fijos se pueden modificar a través del argumento control.fixed=list(...) en la función inla, utilizando siempre los nombres que atribuye INLA a los diferentes parámetros e hiperparámetros (short name). Por ejemplo, si queremos modificar la precisión de los efectos fijos para hacerla igual a 0.001 (esto es, varianza 1000), utilizamos la siguiente sintaxis:
formula = weight ~ 1+ repwt
fit=inla(formula,family="gaussian",data=davis,
control.fixed=list(prec=0.001,prec.intercept=0.001))Volvemos sobre nuestro ejemplo, y asumiendo las a priori por defecto de INLA tendremos: \[\begin{eqnarray*} (y_i|\theta,\beta,\sigma^2) & \sim & N(\theta+\beta x_i,\sigma^2), i=1,...,n \\ \theta & \sim & N(0,\infty) \\ \beta & \sim & N(0,1000) \\ \tau=1/\sigma^2 & \sim & Ga(1,0.00005) \end{eqnarray*}\]
2.6 Resultados
Para mostrar una descriptiva de los resultados del ajuste obtenido con fit=inla(...),
utilizamos la sintaxis siguiente:
-
summary(fit)proporciona una descriptiva del ajuste -
names(fit$marginals.fixed)lista los nombres de todos los efectos fijos -
fit$summary.fixedresume la inferencia posterior sobre los efectos fijos -
names(fit$marginals.hyperpar)lista los nombres de todos los hiperparámetros -
fit$summary.hyperparda un resumen de la inferencia posterior de los parámetros e hiperparámetros -
fit$summary.fitted.valuesresume la inferencia posterior sobre los valores ajustados -
fit$mlikda la estimación de la log-verosimilitud marginal, útil para evaluar y comparar modelos.
Veamos los resultados para nuestro problema de regresión.
# ajuste del modelo
formula = weight ~ 1+ repwt
fit=inla(formula,family="gaussian",data=davis)
summary(fit)
#>
#> Call:
#> c("inla.core(formula = formula, family = family,
#> contrasts = contrasts, ", " data = data, quantiles =
#> quantiles, E = E, offset = offset, ", " scale =
#> scale, weights = weights, Ntrials = Ntrials, strata =
#> strata, ", " lp.scale = lp.scale, link.covariates =
#> link.covariates, verbose = verbose, ", " lincomb =
#> lincomb, selection = selection, control.compute =
#> control.compute, ", " control.predictor =
#> control.predictor, control.family = control.family,
#> ", " control.inla = control.inla, control.fixed =
#> control.fixed, ", " control.mode = control.mode,
#> control.expert = control.expert, ", " control.hazard
#> = control.hazard, control.lincomb = control.lincomb,
#> ", " control.update = control.update,
#> control.lp.scale = control.lp.scale, ", "
#> control.pardiso = control.pardiso, only.hyperparam =
#> only.hyperparam, ", " inla.call = inla.call, inla.arg
#> = inla.arg, num.threads = num.threads, ", "
#> blas.num.threads = blas.num.threads, keep = keep,
#> working.directory = working.directory, ", " silent =
#> silent, inla.mode = inla.mode, safe = FALSE, debug =
#> debug, ", " .parent.frame = .parent.frame)")
#> Time used:
#> Pre = 2.41, Running = 0.192, Post = 0.019, Total = 2.62
#> Fixed effects:
#> mean sd 0.025quant 0.5quant 0.975quant mode
#> (Intercept) 2.734 0.814 1.135 2.734 4.333 NA
#> repwt 0.958 0.012 0.935 0.958 0.982 NA
#> kld
#> (Intercept) 0
#> repwt 0
#>
#> Model hyperparameters:
#> mean sd
#> Precision for the Gaussian observations 0.199 0.021
#> 0.025quant 0.5quant
#> Precision for the Gaussian observations 0.161 0.198
#> 0.975quant mode
#> Precision for the Gaussian observations 0.242 NA
#>
#> Marginal log-Likelihood: -426.73
#> is computed
#> Posterior summaries for the linear predictor and the fitted values are computed
#> (Posterior marginals needs also 'control.compute=list(return.marginals.predictor=TRUE)')
fit$mlik
#> [,1]
#> log marginal-likelihood (integration) -426.7385
#> log marginal-likelihood (Gaussian) -426.7323Obtenemos pues la salida con un descriptivo de la distribución posterior para los efectos fijos interceptación, \(\theta\), y el coeficiente del regresor repwt, \(\beta\), con la media, desviación típica y cuantiles con los que podemos evaluar la región creíble al 95%.
También muestra a continuación una tabla con los descriptivos de la distribución posterior para la precisión \(\tau=1/\sigma^2\) de los datos.
Si queremos obtener los nombres con los que INLA reconoce los efectos fijos (interceptación y coeficiente del regresor) e hiperparámetros (precisión de los datos), llamamos a
names(fit$marginals.fixed)
#> [1] "(Intercept)" "repwt"
names(fit$marginals.hyperpar)
#> [1] "Precision for the Gaussian observations"Y podemos pedir descriptivos específicos de las distribuciones de los efectos fijos y de los hiperparámetros.
# descriptivos efectos fijos
fit$summary.fixed
#> mean sd 0.025quant 0.5quant
#> (Intercept) 2.7338111 0.81438483 1.1348914 2.7338110
#> repwt 0.9583742 0.01213663 0.9345457 0.9583742
#> 0.975quant mode kld
#> (Intercept) 4.3327314 NA 6.303549e-10
#> repwt 0.9822026 NA 6.306970e-10
# descriptivos varianza
fit$summary.hyperpar
#> mean
#> Precision for the Gaussian observations 0.1990956
#> sd
#> Precision for the Gaussian observations 0.02088519
#> 0.025quant
#> Precision for the Gaussian observations 0.1608475
#> 0.5quant
#> Precision for the Gaussian observations 0.1983642
#> 0.975quant mode
#> Precision for the Gaussian observations 0.2418474 NA
# medias de los efectos fijos
fit$summary.fixed$mean
#> [1] 2.7338111 0.9583742
# descriptivos primer efecto fijo
fit$summary.fixed[1,]
#> mean sd 0.025quant 0.5quant
#> (Intercept) 2.733811 0.8143848 1.134891 2.733811
#> 0.975quant mode kld
#> (Intercept) 4.332731 NA 6.303549e-10Para describir la marginal posterior sobre cada uno de los datos ajustados:
head(fit$summary.fitted.values)
#> mean sd 0.025quant 0.5quant
#> fitted.Predictor.001 76.52862 0.2162698 76.10405 76.52862
#> fitted.Predictor.002 51.61089 0.2441494 51.13159 51.61089
#> fitted.Predictor.003 54.48602 0.2190068 54.05607 54.48602
#> fitted.Predictor.004 69.82000 0.1750354 69.47638 69.82000
#> fitted.Predictor.005 59.27789 0.1856012 58.91352 59.27789
#> fitted.Predictor.006 75.57025 0.2087672 75.16040 75.57025
#> 0.975quant mode
#> fitted.Predictor.001 76.95319 NA
#> fitted.Predictor.002 52.09020 NA
#> fitted.Predictor.003 54.91596 NA
#> fitted.Predictor.004 70.16362 NA
#> fitted.Predictor.005 59.64225 NA
#> fitted.Predictor.006 75.98009 NASi queremos hacer un análisis de sensibilidad sobre las distribuciones a priori, reajustamos el modelo con otras priors y comparamos los resultados.
# ajuste del modelo
formula = weight ~ 1+ repwt
fit2=inla(formula,family="gaussian",data=davis,
control.fixed = list(mean.intercept = 100,
prec.intercept = 0.001,
prec = 0.001),
control.family = list(hyper = list(
prec = list(prior="loggamma", param =c(1,0.001)))))
fit2$summary.fixed
#> mean sd 0.025quant 0.5quant
#> (Intercept) 2.7982510 0.81395827 1.2011565 2.7979109
#> repwt 0.9574342 0.01213043 0.9336043 0.9574392
#> 0.975quant mode kld
#> (Intercept) 4.397273 NA 6.167716e-10
#> repwt 0.981236 NA 6.162515e-10
fit2$summary.hyperpar
#> mean
#> Precision for the Gaussian observations 0.1990551
#> sd
#> Precision for the Gaussian observations 0.02087866
#> 0.025quant
#> Precision for the Gaussian observations 0.1603498
#> 0.5quant
#> Precision for the Gaussian observations 0.1983172
#> 0.975quant mode
#> Precision for the Gaussian observations 0.2418039 NA2.7 Distribuciones posteriores
Para obtener la distribución marginal de los valores ajustados y predichos necesitamos incorporar a la función inla el argumento control.compute=list(return.marginals.predictor=TRUE).
Tras conseguir un ajuste con inla, podemos acceder a todas las distribuciones marginales posteriores y predictivas a través de:
-
fit$marginals.fixedda las distribuciones posteriores marginales de los efectos fijos -
fit$marginals.fixed$xxda la distribución del efecto fijoxx, y también se puede seleccionar con su ordinal en el conjunto de efectos fijosfit$marginals.fixed[[i]] -
fit.marginals.hyperparda las distribuciones posteriores marginales de los parámetros e hiperparámetros -
fit$marginals.fitted.valuesda las distribuciones posteriores marginales para los valores ajustados
Con estas distribuciones, reconocidas como marginal, podemos hacer cálculos y gráficos de interés a través de estas funciones que operan sobre las distribuciones y que podemos consultar con ?inla.marginal:
-
inla.dmarginal(x, marginal, ...)da la densidad en x -
inla.pmarginal(q, marginal, ...)da las probabilidades o función de distribución -
inla.qmarginal(p, marginal,...)da los cuantiles -
inla.rmarginal(n, marginal)permite obtener \(n\) simulaciones -
inla.hpdmarginal(p, marginal,...)da la región HPD -
inla.smarginal(marginal, ...)da un suavizado con splines de la distribución marginal -
inla.emarginal(fun, marginal, ...)calcula el valor esperado de una función -
inla.mmarginal(marginal,...)calcula la moda posterior -
inla.tmarginal(fun, marginal,...)transforma la distribución marginal de una función del parámetro -
inla.zmarginal(marginal,...)calcula descriptivos de la marginal.
Veamos algunos ejemplos sobre nuestro problema. Vamos a mostrar a continuación, en un único gráfico, las distribuciones posteriores de los efectos fijos y el parámetro de dispersión de los datos $, con líneas verticales que marquen el valor esperado posterior (en azul) y el HPD95% en rojo.
# library(gridExtra)
# library(ggplot2)
g=list() # lista en que almacenamos los gráficos con d.posteriores
# Efectos fijos
names.fixed=names(fit$marginals.fixed)
n.fixed=length(names.fixed)
names=c(expression(theta),expression(beta))
for (i in 1:n.fixed){
g[[i]] = ggplot(as.data.frame(fit$marginals.fixed[[i]])) +
geom_line(aes(x = x, y = y)) +
labs(x=names[i],y="")+
geom_vline(xintercept=inla.hpdmarginal(0.95,fit$marginals.fixed[[i]]),
linetype="dashed",color="red")+
geom_vline(xintercept=inla.emarginal(function(x) x,fit$marginals.fixed[[i]]),
linetype="dashed",color="blue")
}
# Parámetros
g[[3]]= ggplot(as.data.frame(fit$marginals.hyperpar[[1]])) +
geom_line(aes(x = x, y = y)) +
labs(x=expression(tau),y="")+
geom_vline(xintercept=inla.hpdmarginal(0.95,fit$marginals.hyperpar[[1]]),
linetype="dashed",color="red")+
geom_vline(xintercept=inla.emarginal(function(x) x,fit$marginals.hyperpar[[1]]),
linetype="dashed",color="blue")
# Transformamos la posterior en tau para obtener la posterior de sigma
sigma.post=inla.tmarginal(function(tau) tau^(-1/2),
fit$marginals.hyperpar[[1]])
# y la pintamos
g[[4]]= ggplot(as.data.frame(sigma.post)) +
geom_line(aes(x = x, y = y)) +
labs(x=expression(sigma),y="")+
geom_vline(xintercept=inla.hpdmarginal(0.95,sigma.post),
linetype="dashed",color="red")+
geom_vline(xintercept=inla.emarginal(function(x) x,sigma.post),
linetype="dashed",color="blue")
library(gridExtra)
grid.arrange(g[[1]],g[[2]],g[[3]],g[[4]],ncol=2)
Si queremos la distribución posterior de alguna de las medias \(\mu_i=\theta+ \beta x_i\), necesitamos añadir el argumento control.compute=list(return.marginals.predictor=TRUE) en inla. Así podremos representar, por ejemplo, la distribución posterior sobre el peso esperado para el individuo que aparece en el registro 1 de la base de datos, \(\mu_1=\theta+\beta x_1\):
fit=inla(formula,family="gaussian",data=davis,
control.compute=list(return.marginals.predictor=TRUE))
ggplot(as.data.frame(fit$marginals.fitted.values[[1]]),aes(x=x,y=y))+
geom_line()+
labs(x=expression(mu[1]),y="D.Posterior")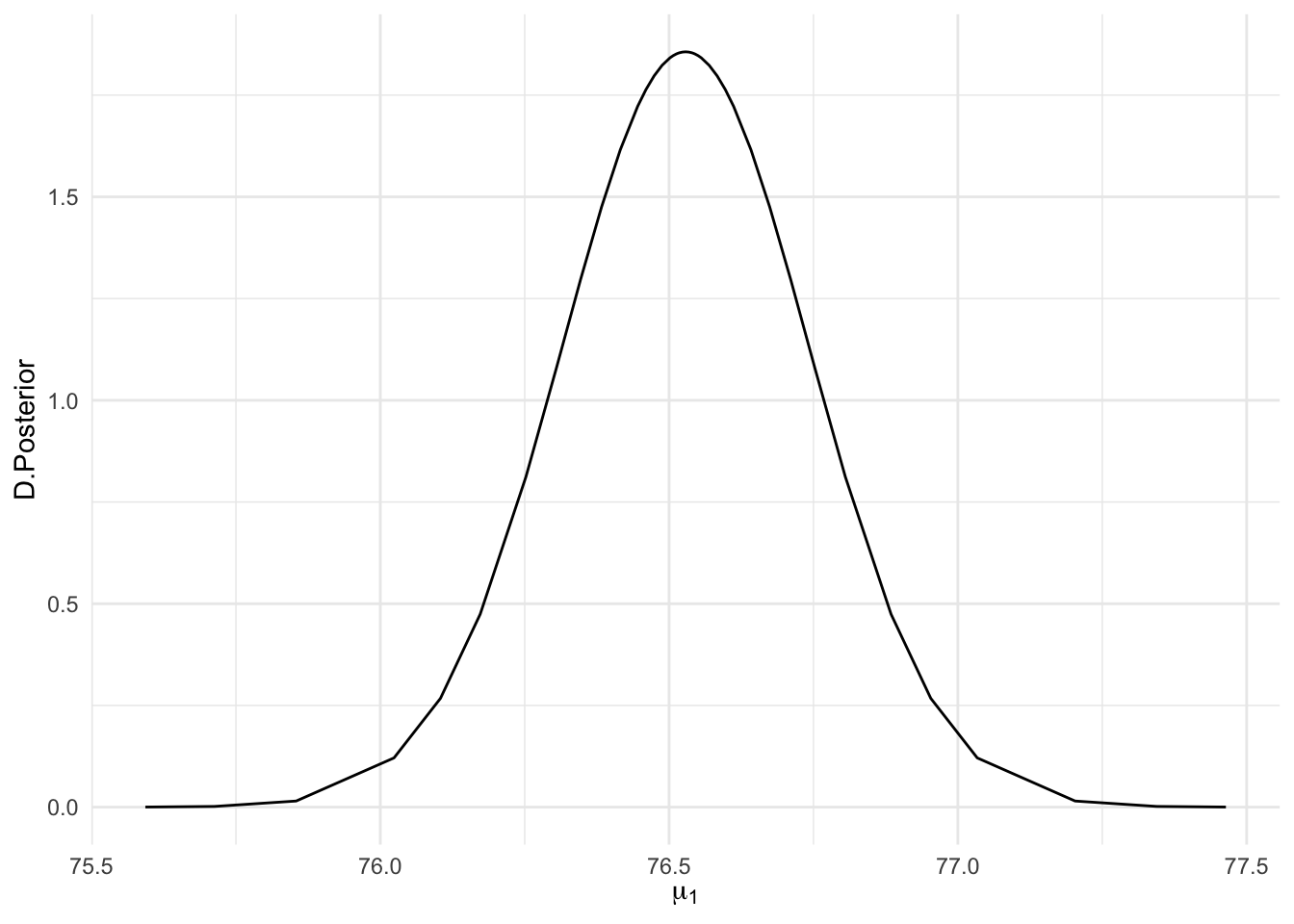
2.8 Simulación de la posterior
Cuando queremos inferir sobre funciones de los efectos latentes e hiperparámetros que no proporciona INLA por defecto , podemos recurrir a simular de las distribuciones posteriores de los efectos involucrados, y con ellas evaluar la función que nos interesa, para conseguir una muestra de su distribución posterior.
Para ello es preciso que al ajustar el modelo con inla hayamos incluido el argumento control.compute=list(config=TRUE).
Para obtener simulaciones de las correspondientes distribuciones posteriores, utilizamos las funciones:
-
inla.posterior.sample(n, fit,selection), para simular los efectos latentes, donde \(n\) es el número de simulaciones pretendido,fites el ajuste obtenido coninlayselectiones una lista con el nombre de las componentes (efectos latentes) a simular.
-
inla.hyperpar.sample(n,fit,improve.marginals=TRUE)para simular de parámetros e hiperparámetros.
Para describir las distribuciones de los nuevos parámetros que queremos evaluar con dichas simulaciones, utilizaremos:
-
inla.posterior.eval()para funciones sobre los efectos fijos -
inla.hyperpar.eval()para funciones sobre los hiperparámetros
Imaginemos que queremos obtener la distribución posterior de \((\theta+\beta \cdot 50)\), que correspondería con el peso real de un sujeto que ha declarado un peso de 50kg. Hemos de simular pues, de las distribuciones posteriores de \(\theta\) y de \(\beta\), para luego aplicar la función correspondiente sobre las simulaciones y obtener una muestra posterior de \(\theta+\beta\cdot 50\).
# reajustamos para poder simular de las posterioris
fit <- inla(formula, data = davis,
control.compute = list(config = TRUE))
# simulamos especificando los parámetros en los que tenemos interés
sims = inla.posterior.sample(100, fit, selection = list("(Intercept)"=1,repwt = 1))Esto nos devuelve una lista de la dimensión del número de simulaciones (cada simulación en un elemento de la lista), y en cada uno de los elementos tenemos los valores simulados de los hiperparámetros (hyper), de los efectos latentes (latent)
length(sims)
#> [1] 100
names(sims[[1]])
#> [1] "hyperpar" "latent" "logdens"
sims[[1]]$hyperpar
#> Precision for the Gaussian observations
#> 0.2507863
sims[[1]]$latent
#> sample:1
#> (Intercept):1 2.0338024
#> repwt:1 0.9690668y la log-densidad de la posterior en esos valores (logdens)
sims[[1]]$logdens
#> $hyperpar
#> [1] 0.2088468
#>
#> $latent
#> [1] 1014.145
#>
#> $joint
#> [1] 1014.354Ahora con la función inla.posterior.sample.eval, dado que nuestra función depende de efectos fijos (latentes), \((\theta,\beta)\), evaluamos la operación pretendida, y con descriptivos gráficos y numéricos de las simulaciones, podemos aproximar los descriptivos de la distribución posterior.
peso_real=inla.posterior.sample.eval(function(...) {(Intercept)+repwt*50},sims)
peso_real
#> sample:1 sample:2 sample:3 sample:4 sample:5
#> fun[1] 50.48714 50.96914 50.88513 50.55123 50.43433
#> sample:6 sample:7 sample:8 sample:9 sample:10
#> fun[1] 50.95626 50.24634 50.67908 50.79885 50.40503
#> sample:11 sample:12 sample:13 sample:14 sample:15
#> fun[1] 50.85233 50.76262 50.49461 50.12846 50.02194
#> sample:16 sample:17 sample:18 sample:19 sample:20
#> fun[1] 50.71335 50.6657 50.22515 50.53359 50.83593
#> sample:21 sample:22 sample:23 sample:24 sample:25
#> fun[1] 50.81653 50.53385 50.37291 50.78633 50.55807
#> sample:26 sample:27 sample:28 sample:29 sample:30
#> fun[1] 51.11283 50.99547 50.99155 50.53261 50.9319
#> sample:31 sample:32 sample:33 sample:34 sample:35
#> fun[1] 50.60077 50.75911 50.88658 50.96568 50.42115
#> sample:36 sample:37 sample:38 sample:39 sample:40
#> fun[1] 51.25975 50.41521 50.30793 50.61346 50.87203
#> sample:41 sample:42 sample:43 sample:44 sample:45
#> fun[1] 50.58164 50.644 50.48652 50.15448 50.39808
#> sample:46 sample:47 sample:48 sample:49 sample:50
#> fun[1] 51.04667 50.79767 50.36373 50.89159 50.82715
#> sample:51 sample:52 sample:53 sample:54 sample:55
#> fun[1] 50.46695 50.73265 50.47238 49.88365 50.42799
#> sample:56 sample:57 sample:58 sample:59 sample:60
#> fun[1] 50.84092 50.92923 50.89597 50.94903 50.56953
#> sample:61 sample:62 sample:63 sample:64 sample:65
#> fun[1] 51.06785 50.44609 50.40124 50.91221 50.37128
#> sample:66 sample:67 sample:68 sample:69 sample:70
#> fun[1] 50.98535 50.97738 50.76141 50.58924 50.6614
#> sample:71 sample:72 sample:73 sample:74 sample:75
#> fun[1] 50.41653 50.95951 50.81905 50.69168 50.63137
#> sample:76 sample:77 sample:78 sample:79 sample:80
#> fun[1] 50.80738 50.37161 50.52499 50.32938 50.64523
#> sample:81 sample:82 sample:83 sample:84 sample:85
#> fun[1] 50.61906 50.46631 50.31782 50.39405 50.6167
#> sample:86 sample:87 sample:88 sample:89 sample:90
#> fun[1] 50.41176 51.17623 50.90767 50.71781 50.4825
#> sample:91 sample:92 sample:93 sample:94 sample:95
#> fun[1] 50.45913 50.39679 50.9806 50.63767 50.55077
#> sample:96 sample:97 sample:98 sample:99 sample:100
#> fun[1] 50.84225 50.97529 50.53797 50.29124 50.43499
pred=data.frame(peso=as.vector(peso_real))
summary(pred)
#> peso
#> Min. :49.88
#> 1st Qu.:50.43
#> Median :50.63
#> Mean :50.64
#> 3rd Qu.:50.86
#> Max. :51.26
ggplot(pred,aes(x=peso))+
geom_histogram(aes(y=..density..), colour="black", fill="white")+
geom_density(alpha=.2, fill="#80E7F5")
#> `stat_bin()` using `bins = 30`. Pick better value with
#> `binwidth`.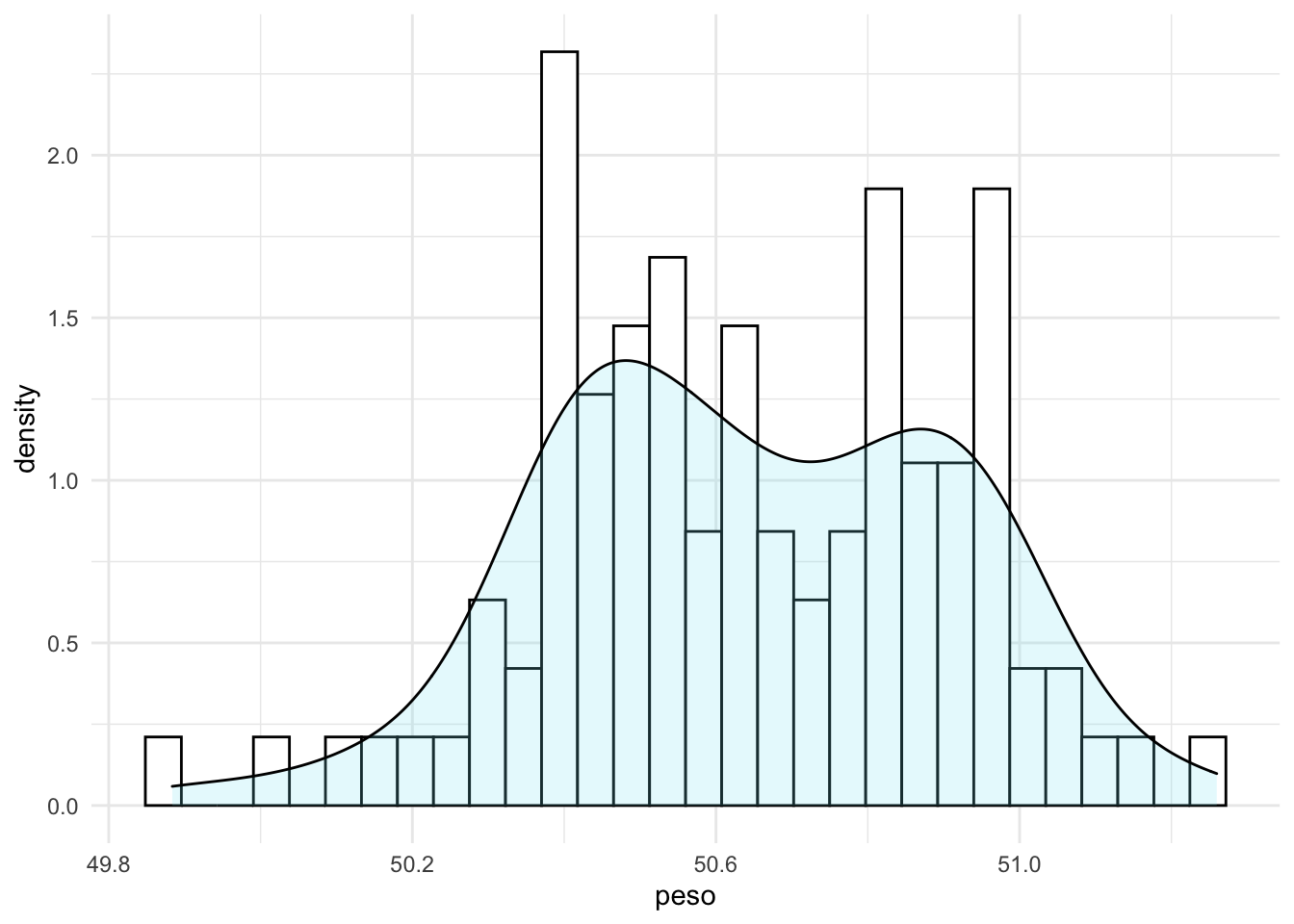
2.9 Regresión lineal múltiple con INLA
Vemos a continuación cómo se trabaja la regresión múltiple con INLA, simplemente añadiendo más predictores.
El modelo de regresión asume una distribución normal para los datos, datos los efectos latentes (todos los relacionados con el valor esperado o predictor lineal) y el resto de parámetros o hiperparámetros del modelo (en nuestro caso la varianza de los datos).
Si tenemos \(n\) observaciones \[ (y_i|\mu_i,\sigma^2) \sim N(\mu_i,\sigma^2), \ \ i=1,...n\] donde \(\mu_i\) representa la media y \(\sigma^2\) la varianza de los datos. \[E(y_i|\mu_i,\sigma^2)=\mu_i, \ Var(y_i|\mu_i,\sigma^2)=\sigma^2\] Si tenemos varios regresores \(x_1,x_2,...,x_J\), la media \(\mu_i\) coincide con el predictor lineal \(\eta_i\) que se construye a partir de una combinación lineal de los predictores: \[\mu_i=\eta_i=\theta+ \sum_{j=1}^J \beta_j x_{ij}\] Nos queda a continuación especificar las distribuciones a priori sobre el vector de efectos latentes, en nuestro caso efectos fijos, \((\theta,\beta_1,...,\beta_J)\), y sobre el parámetro de dispersión o hiperparámetro \(\sigma^2\).
Cuando no tenemos información previa especificamos distribuciones difusas (vagas) sobre los parámetros. En INLA por defecto tendremos:
\[\begin{eqnarray*} \theta &\sim& N(0,\sigma_{\theta}^2) \\ \beta_j &\sim& N(0,\sigma_{\beta}^2) \\ log(\tau=1/\sigma^2) &\sim& Log-Ga(1,0.00005) \end{eqnarray*}\] con \(\sigma_{\theta}^2=\infty\) y \(\sigma_{\beta}^2=1000\).
Ejemplificamos el análisis de regresión lineal múltiple sobre la base de datos usair, en la librería brinla. Esta BD contiene datos recopilados para investigar los factores determinantes de la polución, utilizando el nivel de SO2 como variable dependiente y las restantes como variables explicativas potenciales. Las relaciones entre las variables que incluye se muestra en la Figura 2.1 a continuación.
data(usair, package = "brinla")
library(GGally) # contiene la función de graficado 'ggpairs'
pairs.chart <- ggpairs(usair, lower = list(continuous = "cor"),
upper = list(continuous = "points", combo = "dot"))
ggplot2::theme(axis.text = element_text(size = 6))
#> List of 1
#> $ axis.text:List of 11
#> ..$ family : NULL
#> ..$ face : NULL
#> ..$ colour : NULL
#> ..$ size : num 6
#> ..$ hjust : NULL
#> ..$ vjust : NULL
#> ..$ angle : NULL
#> ..$ lineheight : NULL
#> ..$ margin : NULL
#> ..$ debug : NULL
#> ..$ inherit.blank: logi FALSE
#> ..- attr(*, "class")= chr [1:2] "element_text" "element"
#> - attr(*, "class")= chr [1:2] "theme" "gg"
#> - attr(*, "complete")= logi FALSE
#> - attr(*, "validate")= logi TRUE
pairs.chart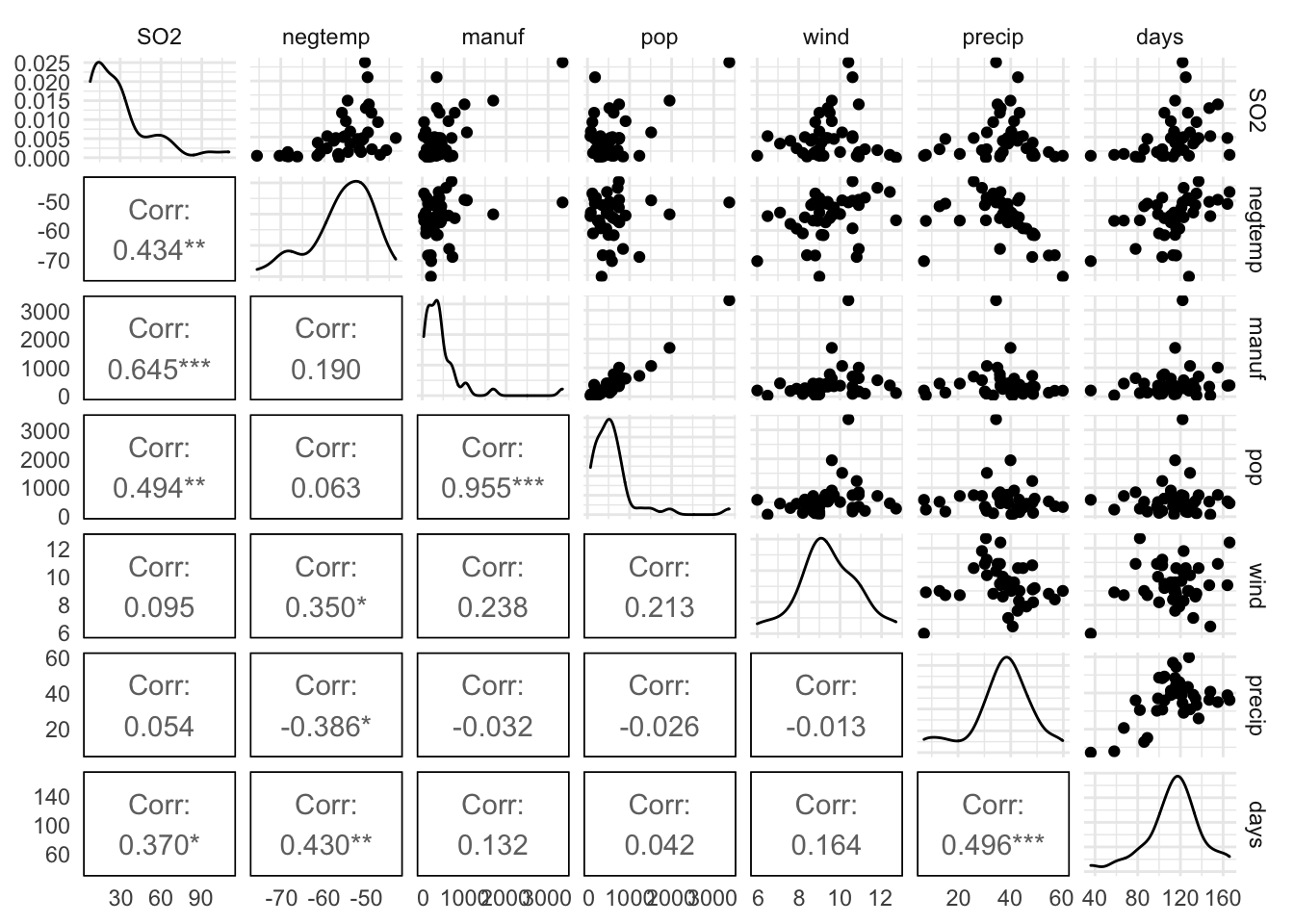
Figura 2.1: Relaciones entre variables en la base de datos usair(brinla).
Apreciamos ya en el gráfico una correlación positiva muy alta entre las variables pop y manuf, y relevante para negtemp y days y también para precip y days, lo que posteriormente condicionará la selección de variables.
2.9.1 Selección de variables
Cuando trabajamos con más de una variable predictora en regresión lineal (realmente en cualquier modelo) surge un problema adicional, que es la selección de variables, o selección del mejor modelo de predicción. Esto se resuelve en INLA utilizando diversos criterios de selección entre los que destacamos:
- la verosimilitud marginal (valor de la log-verosimilitud):
mlik; al cambiarle el signo tendremos -(log-likelihood) - el criterio de información de la deviance (DIC):
dic - el criterio de información bayesiana ampliado (WAIC):
waic - la transformada integral predictiva (PIT):
cpo
El procedimiento a utilizar para la selección del modelo (de variables) es el siguiente:
- ajustar todos los modelos resultantes de todas las combinaciones posibles de predictoras,
- calcular los índices de selección para cada uno de ellos
- proceder con la selección en base a dichos valores.
Siempre se prefieren modelos con los valores más bajos para estos criterios y se descartan los que proporcionan valores más altos. Cuando no es el mismo modelo el que proporciona el valor mínimo en estos criterios, habremos de optar por alguno de ellos.
Por defecto, al ajustar un modelo con inla, nos devuelve la log-verosimilitud marginal (accesible con fit$mlik si el ajuste se guardó en el objeto fit). Para obtener las otras medidas de selección, hemos de incluir como argumento de la función inla, la opción control.compute = list(dic = TRUE, waic = TRUE)). Los valores por defecto de control.compute los podemos consultar ejecutando el comando inla.set.control.compute.default().
Vayamos pues con el ajuste del modelo de regresión con todas las variables. Recordemos que si no especificamos el argumento family, interpreta por defecto la opción gaussian, esto es, normalidad para los datos. Así ajustamos el modelo y obtenemos las siguientes inferencias sobre los efectos fijos.
formula <- SO2 ~ negtemp + manuf + wind + precip + days
fit= inla(formula, data = usair,
control.compute = list(dic = TRUE, waic = TRUE))
#summary(fit)
round(fit$summary.fixed,3)
#> mean sd 0.025quant 0.5quant 0.975quant
#> (Intercept) 135.487 49.861 37.151 135.495 233.780
#> negtemp 1.769 0.634 0.518 1.769 3.020
#> manuf 0.026 0.005 0.017 0.026 0.035
#> wind -3.723 1.935 -7.536 -3.723 0.093
#> precip 0.625 0.387 -0.139 0.625 1.388
#> days -0.057 0.174 -0.400 -0.057 0.287
#> mode kld
#> (Intercept) NA 0
#> negtemp NA 0
#> manuf NA 0
#> wind NA 0
#> precip NA 0
#> days NA 0La inferencia posterior sobre la desviación típica de los datos, \(\sigma\), la resolvemos con sus descriptivos.
sigma.post= inla.tmarginal(function(tau) tau^(-1/2),
fit$marginals.hyperpar[[1]])
inla.zmarginal(sigma.post)
#> Mean 15.6813
#> Stdev 1.84661
#> Quantile 0.025 12.5832
#> Quantile 0.25 14.3546
#> Quantile 0.5 15.4971
#> Quantile 0.75 16.8026
#> Quantile 0.975 19.8219Para seleccionar las variables relevantes seguimos el procedimiento descrito anteriormente. Añadimos también el ajuste del modelo de regresión frecuentista, para el que calculamos como criterio de bondad de ajuste el AIC.
# variables en la bd
vars=names(usair)[-1] # variables predictoras (excluye v.dpte)
nvars=length(vars) # nº variables predictoras
# Truco para concatenar todos los modelos posibles en una fórmula (de Faraway)
listcombo <- unlist(sapply(1:nvars,
function(x) combn(nvars, x, simplify=FALSE)),
recursive=FALSE)
predterms <- lapply(listcombo,
function(x) paste(vars[x],collapse="+"))
nmodels <- length(listcombo)
coefm <- matrix(NA,length(listcombo),4,
dimnames=list(predterms,c("AIC","DIC","WAIC","MLIK")))
# Ajuste de todos los modelos posibles
for(i in 1:nmodels){
formula <- as.formula(paste("SO2 ~ ",predterms[[i]]))
# modelo frecuentista
lmi <- lm(formula, data=usair)
# modelo bayesiano
result <- inla(formula, family="gaussian", data=usair, control.compute=list(dic=TRUE, waic=TRUE))
coefm[i,1] <- AIC(lmi)
coefm[i,2] <- result$dic$dic
coefm[i,3] <- result$waic$waic
coefm[i,4] <- -result$mlik[1]
}Ya solo resta comparar los resultados, respecto de cada uno de los criterios, para seleccionar con qué variables nos quedamos, y por lo tanto con qué modelo de predicción. Basta con encontrar el modelo que proporciona el mínimo valor en cada uno de los criterios.
gana.aic = predterms[which.min(coefm[,1])]
gana.dic = predterms[which.min(coefm[,2])]
gana.waic = predterms[which.min(coefm[,3])]
gana.mlik = predterms[which.min(coefm[,4])]
gana.aic;gana.dic
#> [[1]]
#> [1] "negtemp+manuf+pop+wind+precip"
#> [[1]]
#> [1] "negtemp+manuf+pop+wind+precip"
gana.waic;gana.mlik
#> [[1]]
#> [1] "negtemp+manuf+pop+wind+precip"
#> [[1]]
#> [1] "manuf"Concluimos pues que, tanto el criterio AIC en el modelo de regresión frecuentista, como los criterios DIC y WAIC en el modelo de regresión bayesiano, proporcionan el mejor ajuste. Este mejor modelo incluye como predictores las variables ‘negtemp+manuf+pop+wind+precip’. Reajustamos el modelo con estas variables para derivar las inferencias y predicciones.
Las inferencias sobre los efectos fijos y la precisión de los datos se muestran a continuación.
formula=SO2 ~ negtemp+manuf+pop+wind+precip
fit=inla(formula, family="gaussian", data=usair, control.compute=list(dic=TRUE, waic=TRUE))
fit$summary.fixed
#> mean sd 0.025quant
#> (Intercept) 100.01542077 30.13189996 40.595824939
#> negtemp 1.12026887 0.41418629 0.303552452
#> manuf 0.06489630 0.01548657 0.034362250
#> pop -0.03934768 0.01488029 -0.068687883
#> wind -3.07256361 1.75616870 -6.534354955
#> precip 0.41922312 0.21542102 -0.005537739
#> 0.5quant 0.975quant mode kld
#> (Intercept) 100.01892454 159.41499344 NA 1.154246e-08
#> negtemp 1.12029498 1.93683617 NA 1.157203e-08
#> manuf 0.06489607 0.09543164 NA 1.158379e-08
#> pop -0.03934735 -0.01000932 NA 1.158317e-08
#> wind -3.07281490 0.39066413 NA 1.151620e-08
#> precip 0.41922947 0.84394766 NA 1.158140e-08
fit$summary.hyperpar
#> mean
#> Precision for the Gaussian observations 0.005066401
#> sd
#> Precision for the Gaussian observations 0.001178478
#> 0.025quant
#> Precision for the Gaussian observations 0.00303683
#> 0.5quant
#> Precision for the Gaussian observations 0.00497558
#> 0.975quant mode
#> Precision for the Gaussian observations 0.007614574 NAY en la Figura 2.2 se muestran las distribuciones posteriores de todos los efectos latentes (efectos fijos) en el modelo: interceptación y coeficientes de los regresores.
nfixed=length(fit$names.fixed)
g=list()
for(i in 1:nfixed){
g[[i]]=ggplot(as.data.frame(fit$marginals.fixed[[i]])) +
geom_line(aes(x = x, y = y)) +
labs(x=fit$names.fixed[i],y="")+
geom_vline(xintercept=inla.hpdmarginal(0.95,fit$marginals.fixed[[i]]),
linetype="dashed",color="red")+
geom_vline(xintercept=inla.emarginal(function(x) x,fit$marginals.fixed[[i]]),
linetype="dashed",color="blue")
}
grid.arrange(g[[1]],g[[2]],g[[3]],g[[4]],g[[5]],g[[6]],ncol=2)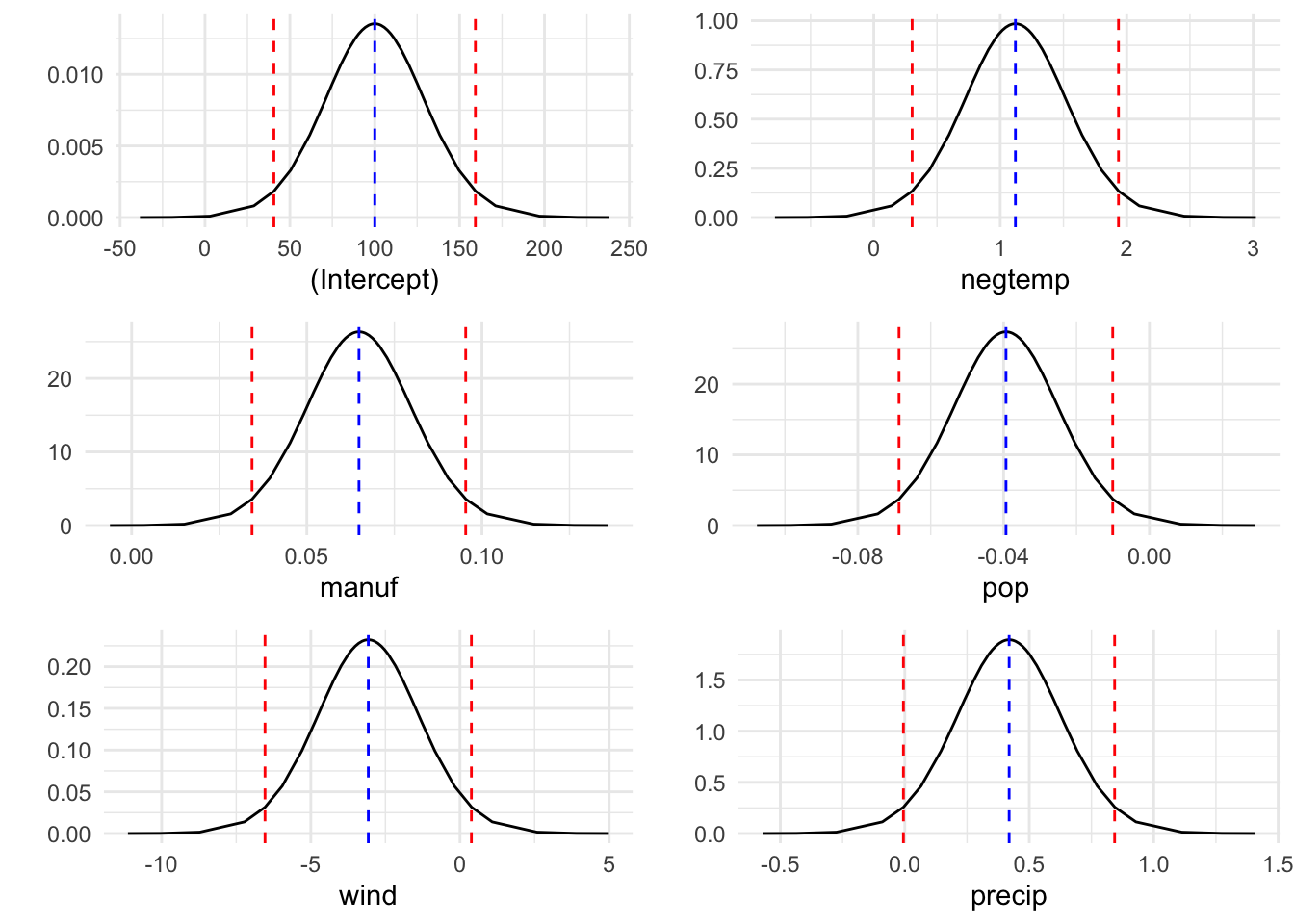
Figura 2.2: Distribuciones posteriores de los efectos latentes.
2.9.2 Predicción de medias
Por último, si queremos predecir la respuesta esperada para ciertos valores de las variables explicativas, no necesariamente existentes en la base de datos, utilizamos el argumento control.predictor en la función inla, especificando en una lista los valores a predecir. Veamos cómo hacerlo con inla, ajustando un modelo sobre un data.frame combinado, en el que añadimos tantas filas como predicciones queremos conseguir, con los valores deseados para los predictores (y valores faltantes en la respuesta), y especificamos los valores a predecir en un vector indicador, a través del argumento control.predictor(list(link=vector.indicador)). Las predicciones se obtienen después, con el resumen de los datos ajustados fitted.values. Recordemos que para poder mostrar las distribuciones predictivas, hemos de añadir en inla el argumento control.compute=list(return.marginals.predictor=TRUE).
# Predicción con INLA
## valores de los predictores en los que predecir: 3 escenarios
formula=SO2 ~ negtemp+manuf+pop+wind+precip
new.data <- data.frame(negtemp = c(-50, -60, -40),
manuf = c(150, 100, 400),
pop = c(200, 100, 300),
wind = c(6, 7, 8),
precip = c(10, 30, 20),
days=c(NA, NA,NA))
## añadimos los tres escenarios de predicción a la bd original,
## dejando como faltantes los valores a predecir de la v.dpte
usair.combinado <- rbind(usair, data.frame(SO2=c(NA,NA,NA),new.data))
## creamos un vector con NA's para observaciones y 1's para predicciones
usair.indicador <- c(rep(NA, nrow(usair)), rep(1, nrow(new.data)))
## reajustamos el modelo añadiendo la opción de predicción de datos
fit.pred <- inla(formula, data = usair.combinado,
control.compute=list(return.marginals.predictor=TRUE),
control.predictor = list(link = usair.indicador))
## y describimos los valores ajustados para los tres escenarios añadidos
fit.pred$summary.fitted.values[(nrow(usair)+1):nrow(usair.combinado),]
#> mean sd 0.025quant 0.5quant
#> fitted.Predictor.42 31.62380 8.203890 15.44856 31.62455
#> fitted.Predictor.43 26.42291 5.473735 15.63094 26.42327
#> fitted.Predictor.44 53.16293 7.091250 39.18167 53.16348
#> 0.975quant mode
#> fitted.Predictor.42 47.79447 NA
#> fitted.Predictor.43 37.21264 NA
#> fitted.Predictor.44 67.14085 NAAsí graficamos en la Figura 2.3 la distribución predictiva del nivel de SO2 para una combinación dada de valores de las variables predictivas, específicamente la que aparece en el escenario 1 propuesto, o lo que es lo mismo, en el registro 42 de la base de datos completada con las nuevas predicciones: negtem=-50, manuf=150, pop=200, wind=6 y precip=10.
pred=fit.pred$marginals.fitted.values[[42]]
ggplot(as.data.frame(pred)) +
geom_line(aes(x = x, y = y)) +
labs(x=expression(eta),y="")+
geom_vline(xintercept=inla.hpdmarginal(0.95,pred),
linetype="dashed",color="red")+
geom_vline(xintercept=inla.emarginal(function(x) x,pred),
linetype="dashed",color="blue")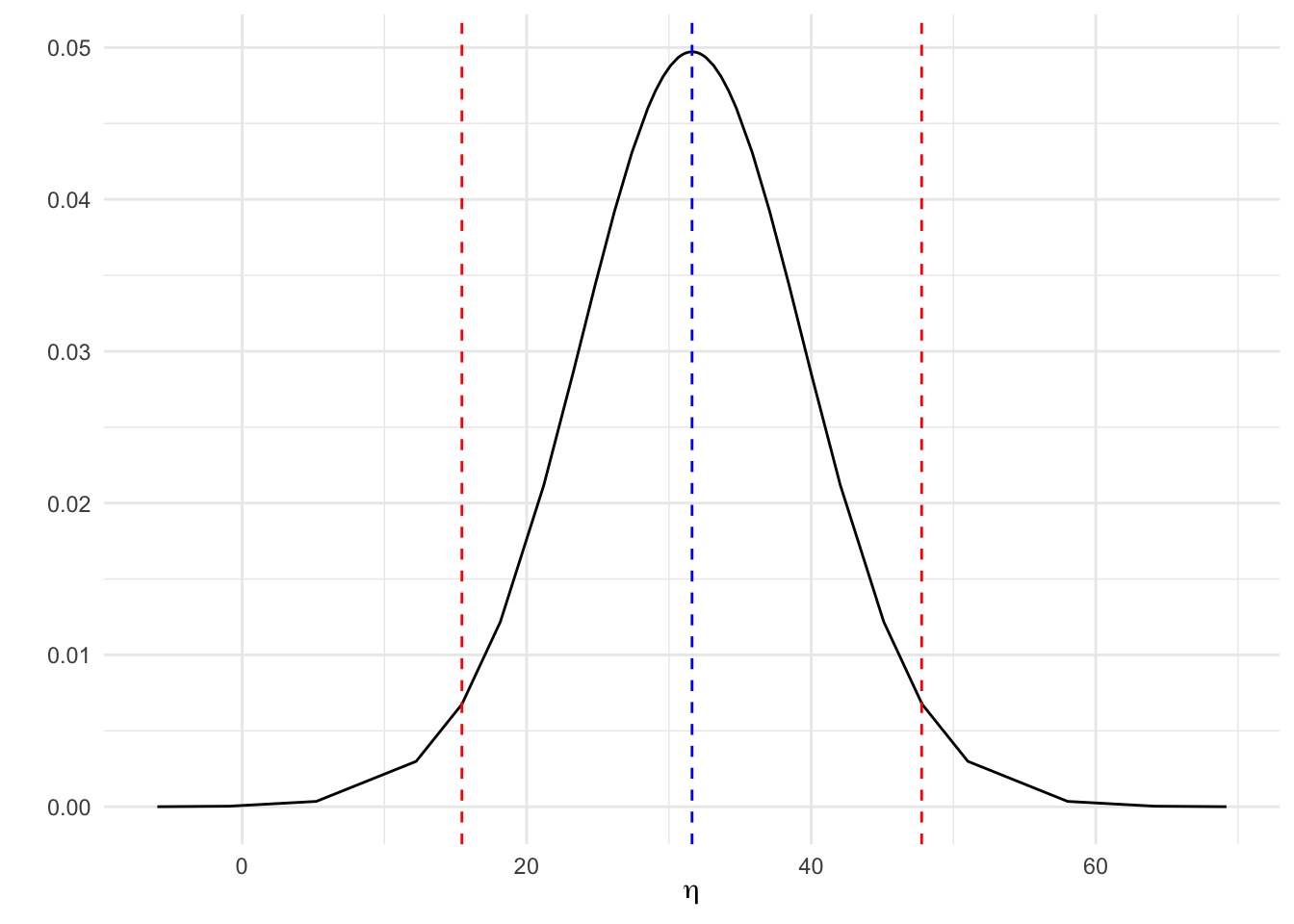
Figura 2.3: Distribución predictiva a posteriori de SO2 para una configuración dada de los predictores.
2.10 Conclusiones
En este tema hemos trabajado con el ajuste con INLA de modelos lineales de regresión, esto es, con efectos fijos. En posteriores temas trabajaremos modelos más sofisticados en los que incluiremos los efectos aleatorios, generalizaremos el modelo lineal y entenderemos el planteamiento de modelos a través de modelos jerárquicos bayesianos.